背景信息
为了将医疗影像用于机器学习算法训练,需要对医疗影像数据进行标注。但是医疗影像中涉及大量的隐私信息,因此医疗影像的打标需要具备私有化部署或离线运行的能力。
图像标注存在不少开源方案,使用最多的是下面这些:
- labelme 11.1k star, 基于 QT 实现可视化
- label-studio 14.3k star, 基于 web 服务可视化,后端使用 Python
- cvat 10.1k star, 基于 web 可视化,后端使用 Python
- labelImg 20.6k star, 基于 QT 实现可视化,并入 label-studio,不再维护了
但是现有这些图像标注工具,基本都没有提供对医疗图像的 DICOM 的支持,因此都无法直接使用。对比了现有的几个项目,最终选择了持续更新的 labelme 项目作为基础,增加对医疗图像的支持,提供开箱即用的医疗图像标注工具 medical-labelme。
labelme 介绍
labelme 是起源于 MIT 的一个开源项目,是一个轻量级的图像标注工具,仅包含图像标注主流程的实现。对加载的图像进行渲染和标注,最终输出 json 格式的标记文件。界面如下所示:
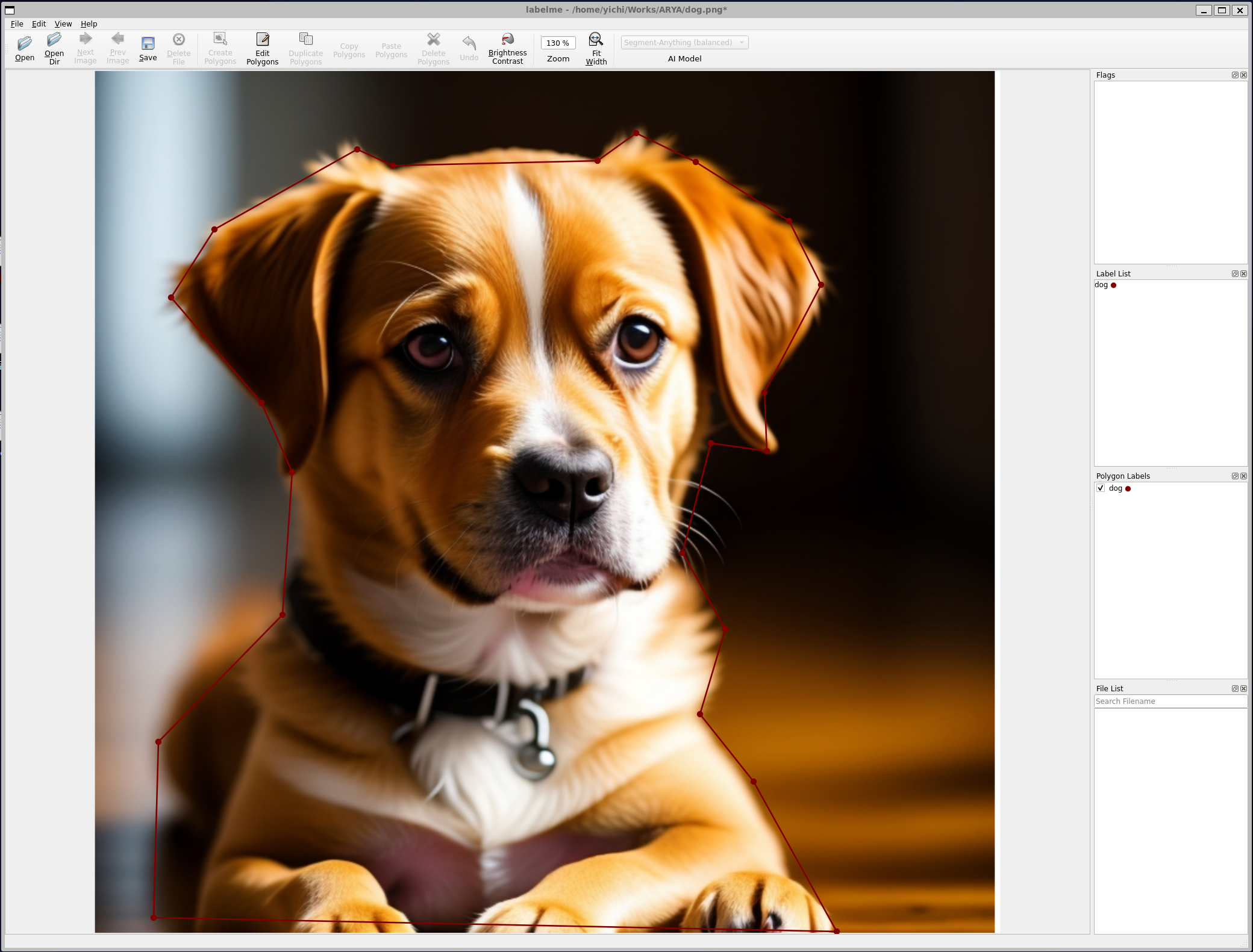
labelme 的主界面实现位于 labelme/app.py,主界面的实现都在 MainWindow 类中。
对应的核心组件为 labelme/widgets/canvas.py,主要实现画布的功能,基于 QPixmap 系统图像组件进行图像展示,并在图像上利用鼠标事件 mousePressEvent 持续确定用户点击的位置,确定用户的标注区域。
medical-labelme 介绍
labelme 对于常规图像的标注已经基本够用,但是缺乏对 DICOM 图像的支持,因此 medical-labelme 尽可能少的改造中实现对医疗图像的支持。改造后效果如下所示:
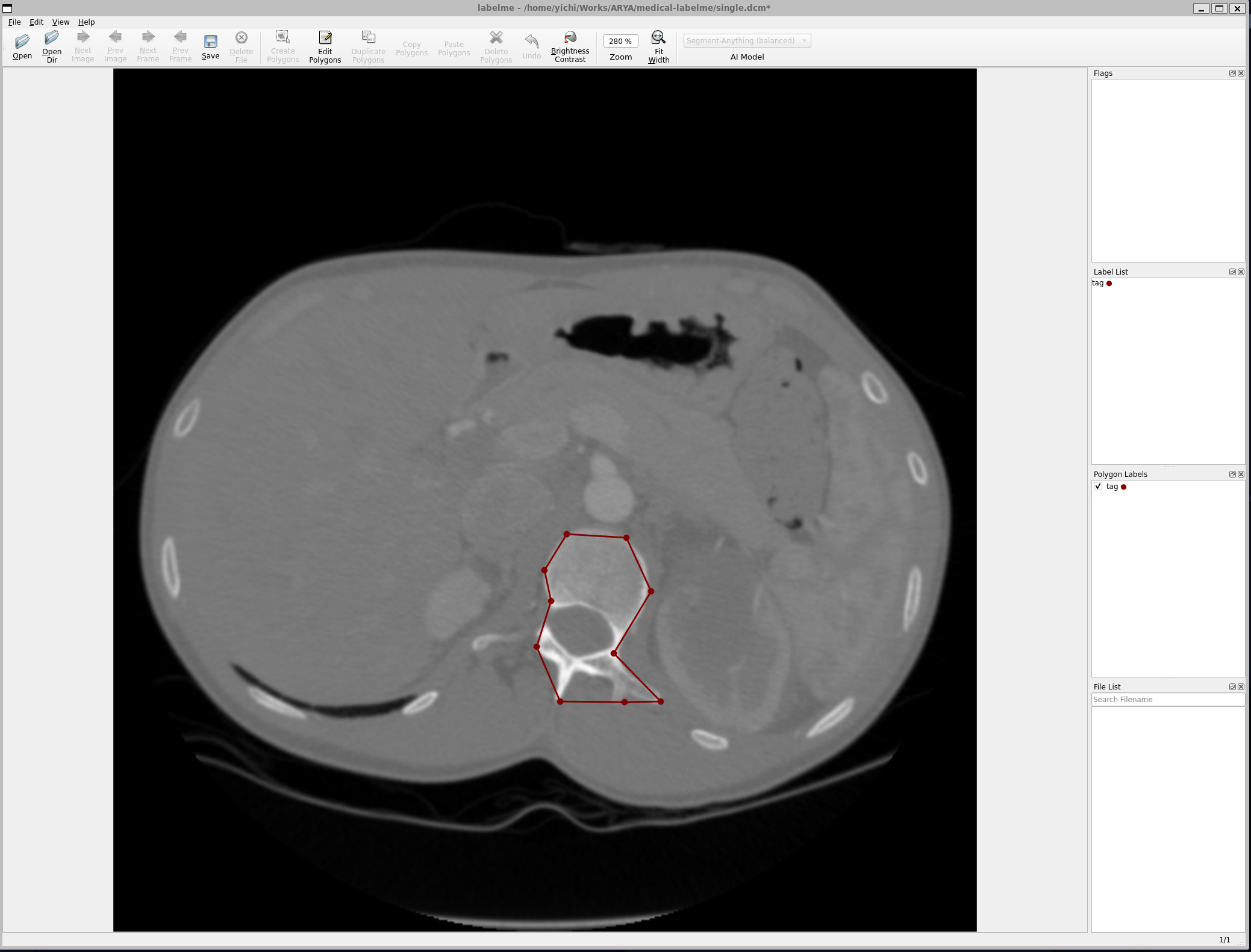
DICOM 解析
在 之前的文章 中对 DICOM 格式与解析进行了简要介绍,在 Python 中主要基于 pydicom 进行图像的解析,使用 pydicom 读取了 DICOM 图像后,可以从 pixel_array 中读取对应的图像数据,此时会得到一个多维的 numpy 列表,一般格式为 帧数 * 宽度 * 高度 * 通道数
此时会碰到的一个明显的问题,常规图像文件一般对应单张图像,而 DICOM 实际可以对应多张图像,此时必然需要额外的处理,这个也是标记 DICOM 图像的一个主要挑战点。
单帧 DICOM 支持
为了扩展 labelme 支持新图像格式,首先需要了解 labelme 是如何支持已有图像格式的,顺着文件加载的流程进行调研,最终确定是通过 labelme/label_file.py 中的 load_image_file()实现的,常规图像的实现比较简单,通过 PIL.image 加载图像,然后保存为字节数据。对应实现如下所示:
def load_image_file(filename):
# 加载图像
image_pil = PIL.Image.open(filename)
image_pil = utils.apply_exif_orientation(image_pil)
# 将原始图像数据保存为特定格式的 IO 字节流
with io.BytesIO() as f:
ext = osp.splitext(filename)[1].lower()
if PY2 and QT4:
format = "PNG"
elif ext in [".jpg", ".jpeg"]:
format = "JPEG"
else:
format = "PNG"
image_pil.save(f, format=format)
f.seek(0)
return f.read()
从上面可以看出,改造主要需要替换掉 image_pil = PIL.Image.open(filename),替换为 DICOM 文件的解析。
DICOM 读取的数据需要做必要的预处理,比如截距 tag 的处理与必要的正则化,才能转换为常规的图像数据,对应的实现如下所示:
def load_dicom_file(filename: str):
# DICOM 文件中处理缩放截距,调整图像数据
def handle_intercept(dicom_data: pydicom.FileDataset,
img_data: np.ndarray):
if "RescaleIntercept" in dicom_data:
img_data += int(dicom_data.RescaleIntercept)
return img_data
# 图像正则化,将原始数据转换为 0 ~ 255 的 rgb 数据
def normalize_img(img_data: np.ndarray):
min_, max_ = float(np.min(img_data)), float(np.max(img_data))
normalize_img_data = (img_data - min_) / (max_ - min_) * 255
trans_img_data = np.uint8(normalize_img_data)
return trans_img_data
# 读取 DICOM 图像
dicom_data = pydicom.dcmread(filename)
img = np.array(dicom_data.pixel_array).astype("float32")
img = handle_intercept(dicom_data, img)
img = normalize_img(img)
image_pil = PIL.Image.fromarray(img)
return image_pil
在原有的 load_image_file() 方法中,判断如果是 DICOM 文件,则调用上面的 load_dicom_file() 进行解析即可。
多帧 DICOM 支持
多帧 DICOM 文件的支持会更复杂一些,因为原始的图像文件只对应一张图片,因此 labelme 的解析 load_image_file() 直接返回图片对应的 IO 字节。而在包含多帧图像的情况下,返回 IO 字节则对应的是多张图的 IO 字节数据,此时无法正确展示与解析。
为了解决这个问题,同时为了尽可能向前兼容,选择了扩展 load_image_file() 方法新增了额外参数 frame, 默认返回第一帧图像,这样返回值与之前就可以保持一致,常规图片直接返回原有数据,多帧 DICOM 图像则实际情况返回对应帧的图像数据。
用户在页面的上切换帧时会传递不同的 frame 参数通过 load_image_file() 获得对应帧的数据。为了保证页面上帧信息的展示,同时为了方面页面上控制用户不会切换超过帧总数,因此 load_image_file() 需要返回总帧数。最终实现改造如下:
def load_dicom_file(filename: str, frame: int = 0):
# 预处理方法 handle_intercept 与 normalize_img 一样
dicom_data = pydicom.dcmread(filename)
img = np.array(dicom_data.pixel_array).astype("float32")
# 获取 dicom 图像对应的全部帧数量,根据 frame 返回对应的帧的数据
total_frame = utils.count_dicom_frame(dicom_data)
if total_frame > 1:
img = img[frame]
img = handle_intercept(dicom_data, img)
img = normalize_img(img)
image_pil = PIL.Image.fromarray(img)
return image_pil, total_frame
def load_image_file(filename: str, frame: int = 0):
total_frame = 0
# 根据类型使用不同的图像解析方式
if utils.is_dicom_file(filename):
image_pil, total_frame = LabelFile.load_dicom_file(filename, frame)
else:
image_pil = LabelFile.load_common_image_file(filename)
total_frame = 1
if not image_pil:
return None, 0
image_pil = utils.apply_exif_orientation(image_pil)
# 将原始图像数据保存为特定格式的 IO 字节流,不需要额外处理,代码与上面一样
实现了上面的能力后,应用层就可以获取到总帧数,方便进行帧信息的展示。并可以通过 frame 参数实现帧切换。为了方便用户切换帧,在页面操作区上增加了帧切换的按钮,在右下角增加了当前帧的展示信息,最终实现的效果如下所示:
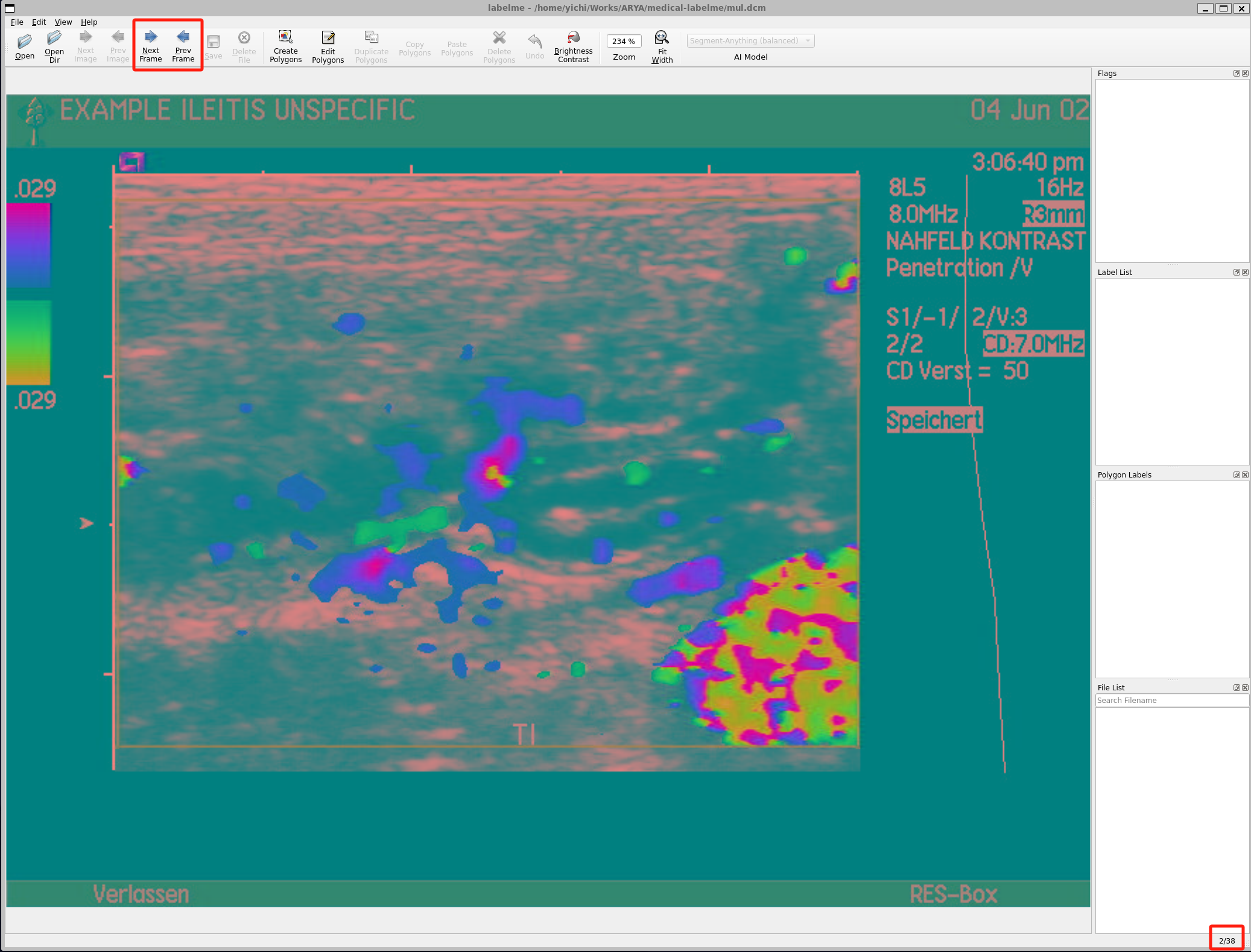
在保存的标注输出文件中,也额外新增了标记帧的字段,这样模型训练时就能选择正确的关键帧了。
总结
通过上面改造,使用尽可能少的开发成本,保证尽可能向前兼容的情况下,增加了对 DICOM 图像数据的支持。但是目前的版本是在很短时间内开发出来的基础版本,虽然基本可用,但是依旧存在不少优化空间,比如帧切换的过程中会多次解析 DICOM 文件,效率不高,预期后续会对 medical-labelme 进行进一步的优化,从而更好地支持 DICOM 图像的标注,有需要的同志可以自行试用。