背景介绍
最近开始推进医疗影像数据机器学习模型,医疗领域最常规的图像格式当属 DICOM 了,之前对医疗图像了解甚少,此次对 DICOM 图像进行了一些探索,整理相关内容在这边。
DICOM 图像探索
DICOM 格式介绍
DICOM 是一组通用标准协议,包括医疗图像的处理,存储,传输等各个方面的内容。协议具体内容可以查看 dicomstandard.org
而之前搜索调研医疗图像最常规的格式是 DICOM 和 NIfTI,实际和医院的科研单位进行沟通,医疗图像使用最多的还是 DICOM 格式,预期后续的医疗图像模型训练主要以 DICOM 为主。
图像结构
DICOM 图像数据主要包含 Header 和 Dataset,其中 Header 是数据头,用于验证 DICOM 格式,Dataset 是实际的数据内容。具体的格式如下所示:
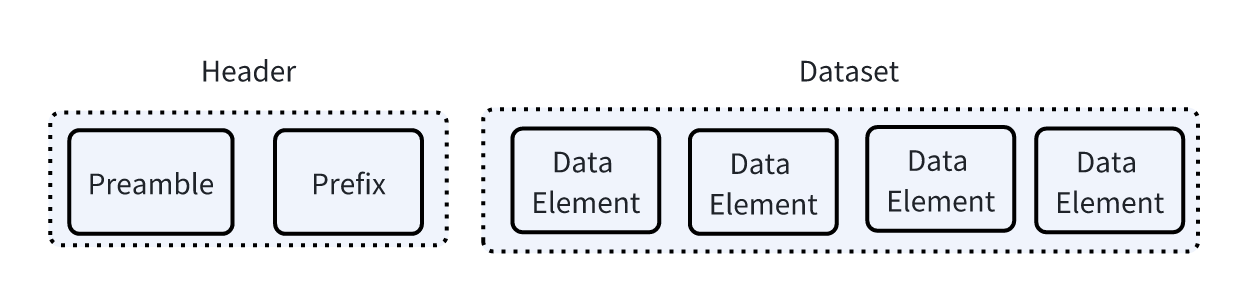
Header
Header 部分主要由两部分组成,Preamble 是 128 字节的填充内容,可以忽略,Prefix 是 DICM 字符。选择一个 DICOM 文件,使用 ImHex 打开查看对应十六进制数据如下:
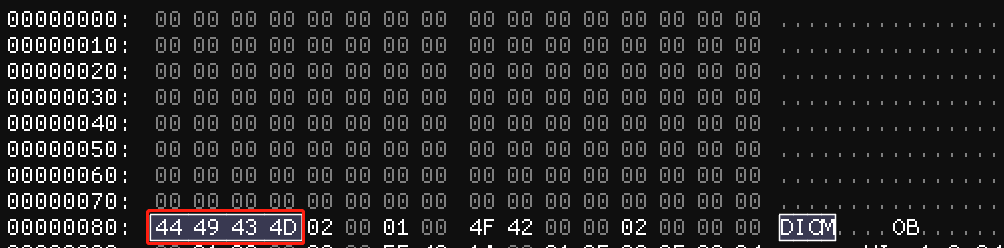
可以看到与具体的描述一致,一般情况下 Header 部分我们都不需要关心,解析工具会基于 Header` 执行必要的校验。
Dataset
Dataset 是以 Data Element 为单位依次排列而成,具体的格式如下所示:
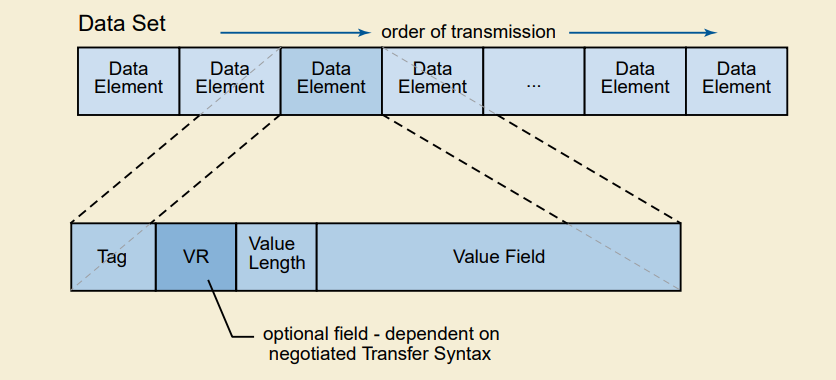
单个 Data Element 主要由四部分组成:
Tag:4 字节,描述数据对应的编号,前面 2 字节为组编号,后面 2 字节为元素编号;VR: 2 字节,可选字段,描述数据对应的类型;Value Length:2 字节,描述数据长度;Value Field:可选长度,实际的数据;
Tag 与 VR 决定了数据的类型,通过这两个字段可以确定对应的数据是病人的姓名,还是设备生产厂家,或者是对应的图像数据,对应关系可以通过 dicomlibrary 进行查询
从这个结构也可以看出,DICOM 文件的特殊之处在于,使用Data Element既支持了描述信息,也支持图像数据,而且 DICOM 可以根据需要被设计为单帧图像或多帧图像。
DICOM 解析
DICOM 图像在不同语言存在不同的解析工具,在 Python 语言中主要使用 pydicom 进行解析。使用方式相对简单:
from pydicom import dcmread
ds = dcmread(file_path)
# 获取图像原始像素数据
print(ds.PixelData)
# 获取图像像素数组
print(ds.pixel_array)
使用 pydicom 解析中,虽然可以通过 PixelData 获取像素数据,但是基本上用处不大,因为 DICOM 文件可能会包含多个图像帧,基于 PixelData 获取到的像素数据就无法表示任一图像。
一般情况下我们会使用 pixel_array 获取像素点 numpy 列表,对于单帧单通道图像,此时获取到的是一个 宽度 * 高度 的二维数组,而多帧多通道图像,此时获取的是 帧数 * 宽度 * 高度 * 通道 的四维列表,通过 numpy 操作可以获取对应帧的图像,甚至可以根据需要进行必要的压缩操作,关于这部分的描述可以参考 pixel data 文档
总结
DICOM 是医疗图像的通用格式,一个特殊设计在于通过 Data Element 列表,支持将描述信息与实际的图像数据使用同样的结构进行描述,可以支持将患者相关信息直接写入 DICOM 图像中,使用更加便利,但是也导致 DICOM 图像本身包含大量敏感隐私信息,因此 DICOM 图片需要被谨慎处理,否则便容易导致信息泄露。